ಟ್ವೀಟ್ಗಳು
- ಟ್ವೀಟ್ಗಳು, ಪ್ರಸ್ತುತ ಪುಟ.
- ಟ್ವೀಟ್ಗಳು & ಪ್ರತಿಕ್ರಿಯೆಗಳು
- ಮಾಧ್ಯಮ
ನೀವು @mikelove ಅವರನ್ನು ತಡೆಹಿಡಿದಿರುವಿರಿ
ಈ ಟ್ವೀಟ್ಗಳನ್ನು ವೀಕ್ಷಿಸಲು ನೀವು ಖಚಿತವಾಗಿ ಬಯಸುವಿರಾ? ಟ್ವೀಟ್ ವೀಕ್ಷಣೆಯು @mikelove ಅವರ ತಡೆತೆರವುಗೊಳಿಸುವುದಿಲ್ಲ
-
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Our new paper: Using Equivalent Class counts directly for testing differential transcript usage. Nice work from
@marekcmero@nadia_davidson https://www.biorxiv.org/content/early/2018/12/19/501106 …pic.twitter.com/JTANHlLQzZ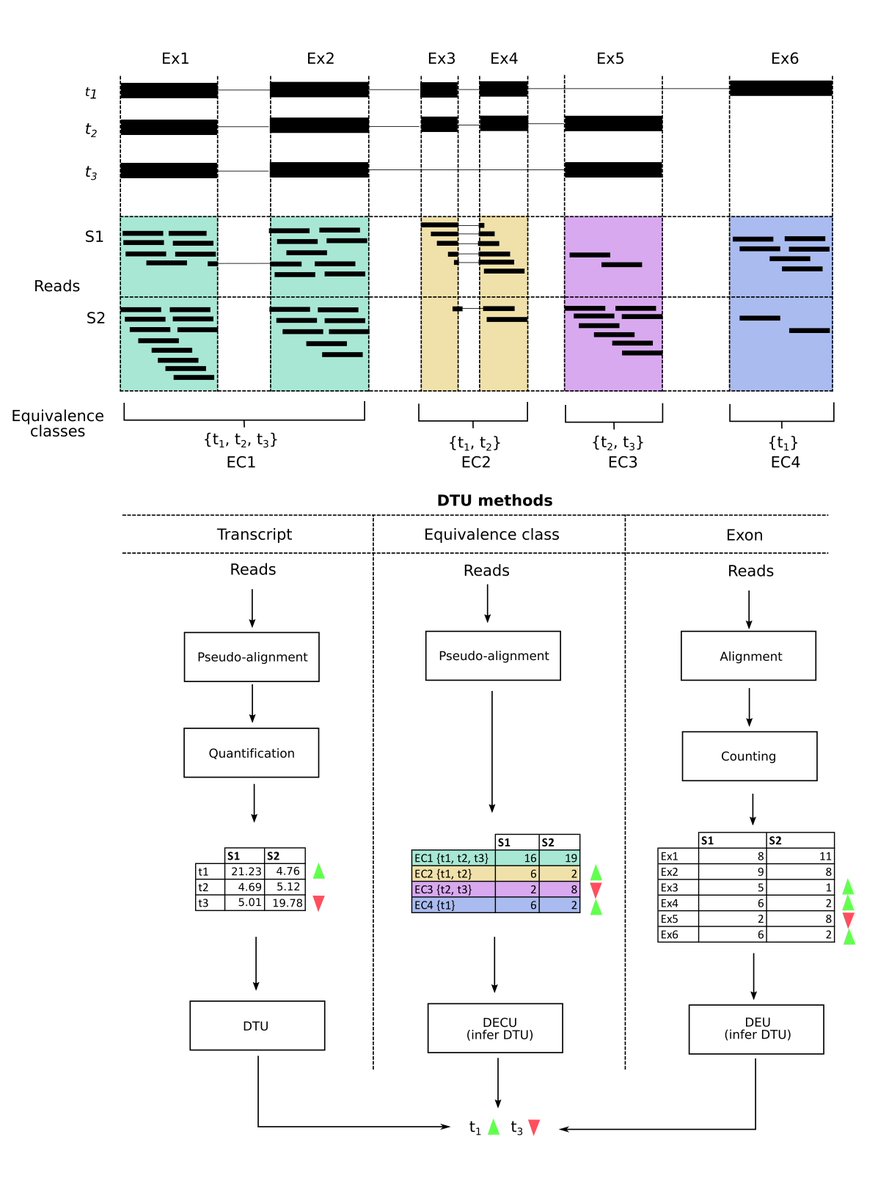 ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
The decontam method introduced in our new
@MicrobiomeJ paper is available as an R package via Bioconductor and has an annotated tutorial. Bioconductor: https://bioconductor.org/packages/release/bioc/html/decontam.html … Tutorial: https://benjjneb.github.io/decontam/vignettes/decontam_intro.html … Manuscript:https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-018-0605-2 …ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Our intensely collaborative (5/6 authors are PIs!) review article *for biologists* about theory / mathematical modeling is out in JCS: Unite to divide – how models and biological experimentation have come together to reveal mechanisms of cytokinesishttp://jcs.biologists.org/content/131/24/jcs203570 …
ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Congrats
@bejcal@hyposalivation and Nicole Davis: really useful piece of work: Simple statistical identification and removal of contaminant sequences in marker-gene and metagenomics datahttps://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-018-0605-2?utm_source=dlvr.it&utm_medium=twitter …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
#rstats PSA: The incoming CRAN submission queue will be closed from Dec 21 to Jan 2 (assumedly for a well deserved break for the volunteer team). Get your new packages and updates in before the 21st or wait until the new year.ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Alevin efficiently estimates accurate gene abundances from dscRNA-seq data https://www.biorxiv.org/content/early/2018/10/24/335000 … I was pleased to see this! Nicely solves an unsightly hack in UMI-based scRNAseq.
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
In stat method life and then in R pkg life, I had to intentionally cultivate a neutral-to-positive attitude re: what others were doing — especially if the work is adjacent to mine. We’re embarked on a collective effort! This makes me much happier and more productive too.
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Our integrative study on dozens of in vivo RNA-binding protein datasets is out! A long standing collaboration with Tom Tuschl and fantastic work led by
@neelmukherj . I think of it as renegade inofficial@ENCODE_NIH companion paper https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gky1185/5230955#.XBarxpEjROE.twitter …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gky1185/5230955#.XBarxpEjROE.twitter …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Very excited to be leading our new Translational
#DataScience Integrated Research Center at@fredhutch So many opportunities in this space bridging many disciplines with terrific computational/experimental investigators. Stay tuned!https://twitter.com/fredhutch/status/1073825736937668608 …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
CancerInSilico: An R/Bioconductor package for combining mathematical and statistical modeling to simulate time course bulk and single cell gene expression data in cancer | bioRxivhttps://www.biorxiv.org/content/early/2018/12/14/328807 …
ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Polygenic risk scores for breast cancer and estrogen-receptor subtypes, developed using individual-level data on 170,000 cases and controls from the Breast Cancer Association Consortium. 313 SNPs can identify 1% of women at 4.3-fold increased risk of ER+ disease.https://twitter.com/AJHGNews/status/1073251358407446529 …
ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
if you use NNLS for deconvolution, you should be adding expression profiles on the raw expression scale, not log scale. we have a helper function unmix() for making comparisons in log-like scale while doing the NNLS on the raw, easy to copy the code and modify
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Breast cancer screening is one of the most tractable uses of
#polygenic risk scoring. Improved predictions, with 32% BC risk in highest 1% PRS. https://www.cell.com/ajhg/fulltext/S0002-9297(18)30405-1#secsectitle0075 …pic.twitter.com/zfTe2PM540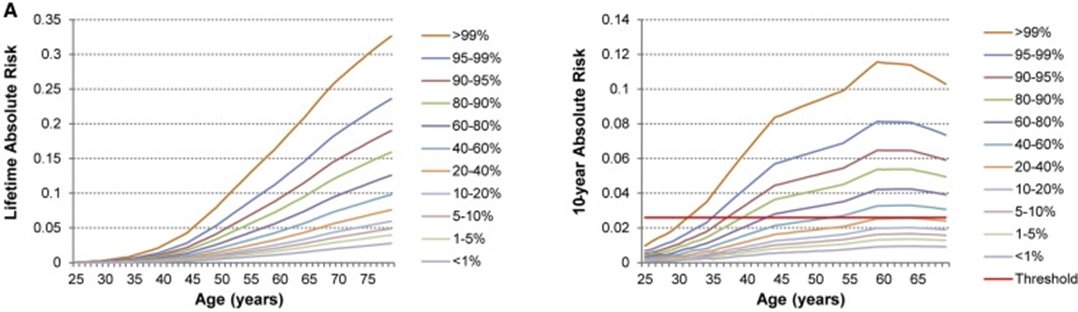 ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Wow. PsychEncode just flooded the science publication group with 10 manuscripts revealing a significant progress made towards understanding the brain's molecular architecture. They dedicate this release to
@PamelaSklar1http://science.sciencemag.org/content/362/6420/1262 …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Random thoughts about confidence intervals: When we teach precision & accuracy, we often use images of a target, like these from the Wikipedia article: https://en.m.wikipedia.org/wiki/Accuracy_and_precision … But I think this analogy leads to confusion about the interpretation of the confidence interval. 1/4pic.twitter.com/8Ma7KxsHjW
 ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು
ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Using multiple measurements of tissue to estimate cell-type-specific gene expression via deconvolutionhttps://www.biorxiv.org/content/early/2018/12/12/379099 …
ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Our study with
@dulaclab on pup recognition just out! Check it out in the following links: https://www.cell.com/cell/fulltext/S0092-8674(18)31557-5 … or https://authors.elsevier.com/a/1YDU1L7PXUjitಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Check out our new ENIGMA GWAS: Common genetic variants impacting the structure of the human cerebral cortex, measured with MRI, in 35k humans, ~200 loci:https://www.biorxiv.org/content/early/2018/09/03/399402 …
ಈ ಥ್ರೆಡ್ ತೋರಿಸಿಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
The Brain Transcriptome maps are finally out! This includes some of the RNA-seq data back from my post-doc in 2008 with
@Genomics_Guy! Revealing the brain's molecular architecture, one transcript at a time - see herehttp://science.sciencemag.org/content/362/6420/1262 …ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು -
Mike Love ಅವರು ಮರುಟ್ವೀಟಿಸಿದ್ದಾರೆ
Important session at
@broadinstitute this morning - a discussion of the need for greater diversity in human genetics, led by@genetisaur. She leads off with a pretty astonishing statistic: Europeans are 80% of samples in genetic studies, vs 16% of the global population.ಧನ್ಯವಾದಗಳು. Twitter ಇದನ್ನು ನಿಮ್ಮ ಕಾಲರೇಖೆಯನ್ನು ಉತ್ತಮಗೊಳಿಸಲು ಬಳಸುತ್ತದೆ. ರದ್ದುಗೊಳಿಸುರದ್ದುಗೊಳಿಸು
ಲೋಡಿಂಗ್ ಸಮಯ ಸ್ವಲ್ಪ ತೆಗೆದುಕೊಳ್ಳುತ್ತಿರುವಂತೆನಿಸುತ್ತದೆ.
Twitter ಸಾಮರ್ಥ್ಯ ಮೀರಿರಬಹುದು ಅಥವಾ ಕ್ಷಣಿಕವಾದ ತೊಂದರೆಯನ್ನು ಅನುಭವಿಸುತ್ತಿರಬಹುದು. ಮತ್ತೆ ಪ್ರಯತ್ನಿಸಿ ಅಥವಾ ಹೆಚ್ಚಿನ ಮಾಹಿತಿಗೆ Twitter ಸ್ಥಿತಿಗೆ ಭೇಟಿ ನೀಡಿ.